项目名称:Chip-seq分析流程
所属分类:生物信息学分析
联系电话:020-85625352
QQ:386244141
Email:servers@gzscbio.com
Chip-seq分析流程
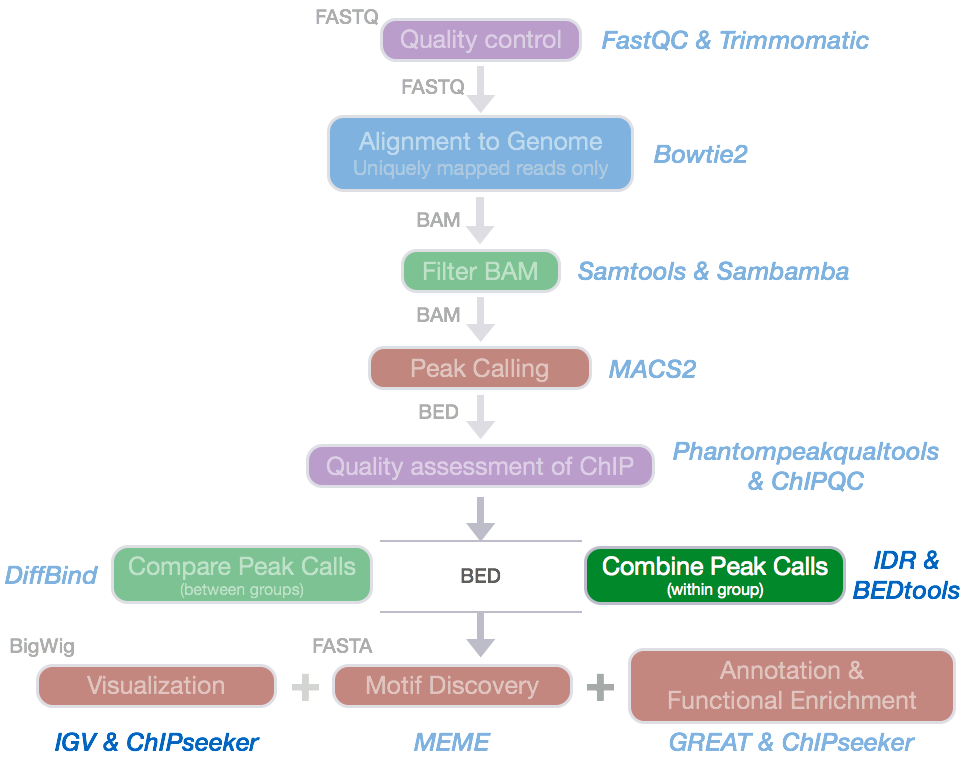
流程的一些关键点分析:
我们的Peak是如何找出来的?Callpeak的流程(MACS2)
1. 质控 (quality control)
首先要看一下ChIP-seq数据的质量,数据的信号最好比background要强很很多。一般要有control,这样call peaks更准确可信, control主要有Input DNA 和 IgG两种,前一种更常用。
检测质量的一些方式:
1). peaks中reads的数量,如果peaks的reads普遍较少,则质量一般。
2). peaks信号高,背景低。
3). 测序深度深 。
4). Diverse library (与重复duplications有关,如下图)
5). 有重复并且与重复之间相似性较高…
……

2. 序列比对 (mapping of fastq)
序列比对一般用BWA或者Bowtie2,两者效果差不多。我们一般采用Bowtie2,对reads进行基因组进行回帖。
3. 去除重复 (remove duplicates)
由于PCR实验存在不可避免的实验误差,所以会存在重复 (duplicates)。我们一般在Chip-seq中会进行去除。
理论上来讲,不同的序列在进行PCR扩增时,扩增的倍数应该是相同的。但是由于聚合酶的偏好性,PCR扩增次数过多的情况下,会导致一些序列持续扩增,而另一些序列扩增到一定程度后便不再进行,也就是我们常说的PCR偏好性。
这种情况对于定量分析(如ChIP-seq),会造成严重的影响。此外,PCR扩增循环数过多,会出现一些扩增偏差,进而影响后续分析结果的置信度。
4. peak calling
peaks是reads信号比较强的区域,也就是我们找到的转录因子或者组蛋白修饰最有可能结合的地方。call peaks仍然有不少软件,比较常用的是MACS2和Hotspot2。
5. 下游分析 (downstream analysis)
分析完之后下游可以做的事情很多,视情况而定。 可分析Peak的临近注释基因,分布类型情况,及功能注释情况; 或者Homer等工具注释peaks,看不同转录因子/组蛋白修饰之间的关系,或者分析TF的target gene。 或者同时分析RNA-seq、ATAC-seq等数据,看转录因子与染色质开放区的关系;