基因表达调控主要实验技术
ChIP、RIP、RNA pull-down、EMSA、Luciferase是基因表达调控研究中,最为核心、最为关键的实验技术。

图1 核心实验(ChIP、RIP、RNA pull-down)原理示意
1 ChIP实验
通过与染色质片段共沉淀和PCR技术,在体内检测与特异蛋白质结合的DNA片段。将处于适当生长时期的活细胞用甲醛交联后将细胞裂解, 染色体分离并打碎为一定大小的片段200bp-1000bp;然后用特异性抗体免疫沉淀目标蛋白与 DNA交联的复合物, 对特定靶蛋白与DNA片段进行富集。采用低pH值条件反交联, DNA与蛋白质之间的 Schiff键水解, 释放DNA片段。通过对目标片段的纯化与检测,获得DNA与蛋白质相互作用的序列信息。
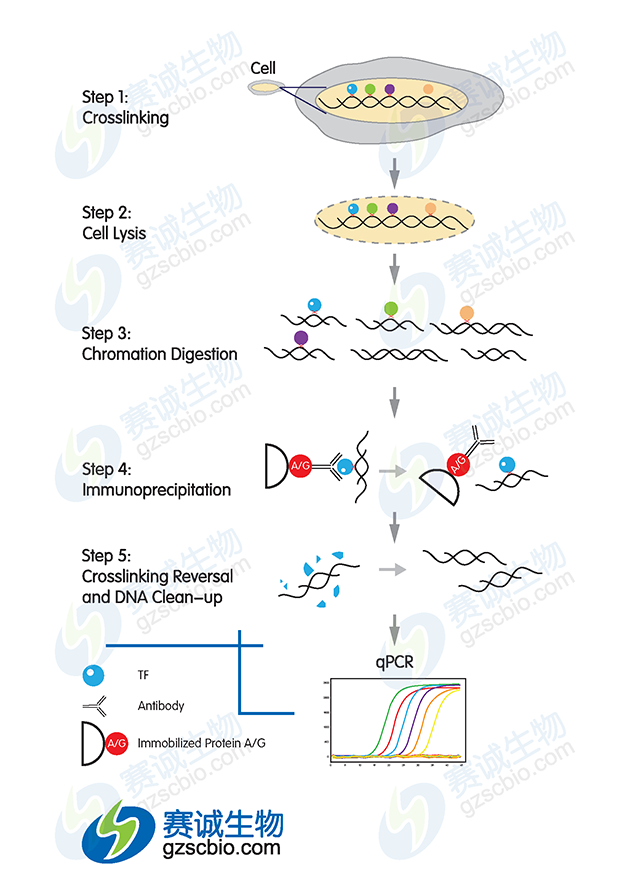
图2 ChIP实验流程
2 RIP实验
RIP技术(RNA Binding Protein Immunoprecipitation,RNA结合蛋白免疫沉淀),是研究细胞内RNA与蛋白结合情况的技术。运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上的RNA进行分析;即用抗体或表位标记物捕获细胞核内或细胞质中内源性的RNA结合蛋白,防止非特异性的RNA的结合,免疫沉淀把RNA结合蛋白及其结合的RNA一起分离出来,结合的RNA序列通过microarray(RIP-Chip),定量RT-PCR或 高通量测序(RIP-Seq)方法来鉴定。是了解转录后调控网络动态过程的有力工具,能帮助我们发现miRNA的调节靶点。
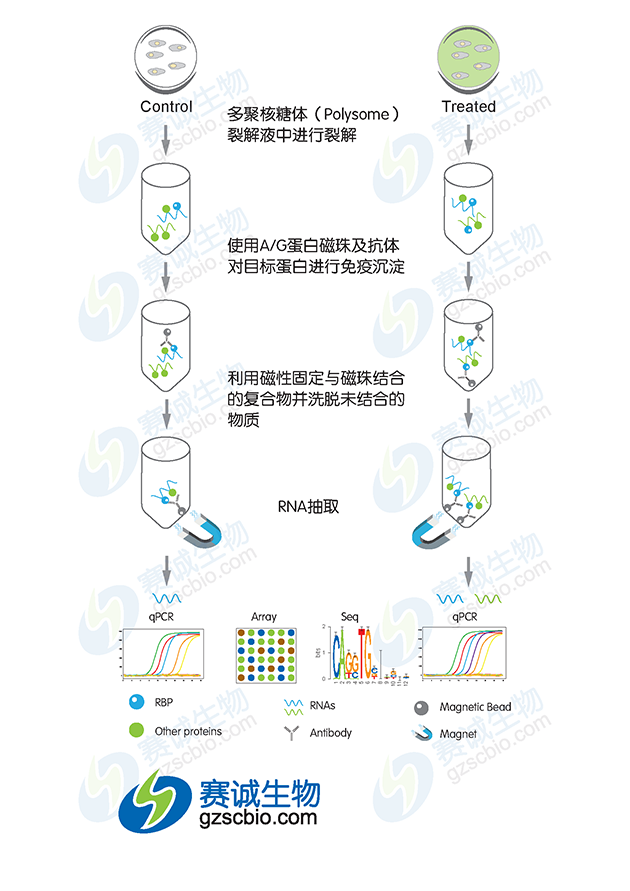
图3 RIP实验流程
3 RNA pull-down实验
使用体外转录法标记生物素RNA探针,然后与胞浆蛋白提取液孵育,形成RNA-蛋白质复合物。该复合物可与链霉亲和素标记的磁珠结合,从而与孵育液中的其他成分分离。复合物洗脱后,通过western blot实验检测特定的RNA结合蛋白是否与RNA相互作用。
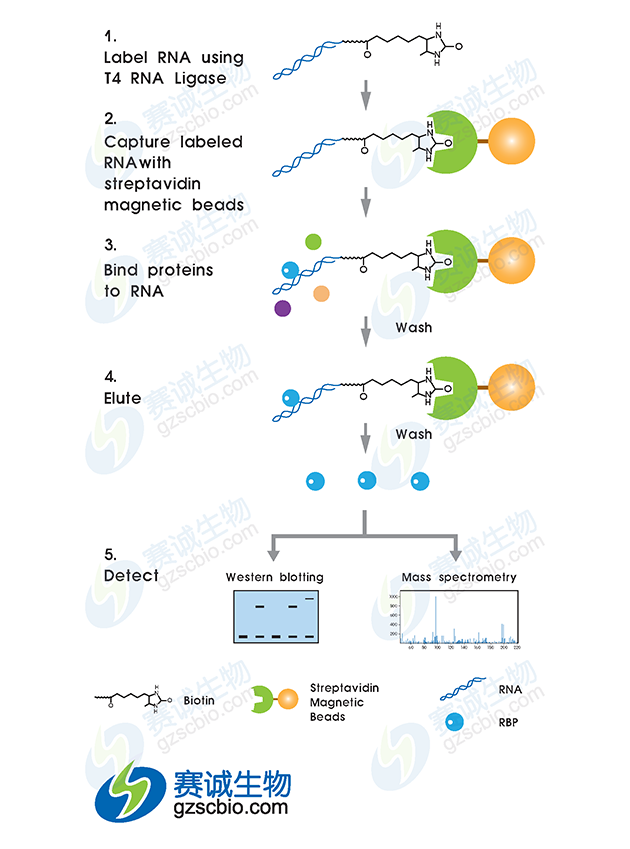
图4 RNA pull-down实验流程
4 EMSA实验
凝胶迁移或电泳迁移率实验(EMSA-electrophoretic mobility shift assay)是一种研究DNA结合蛋白和其相关的DNA结合序列相互作用的技术,可用于研究DNA结合蛋白和其相关的DNA结合序列相互作用、DNA定性和定量分析。生物素标记的探针依研究的结合蛋白的不同,可是双链或者是单链。
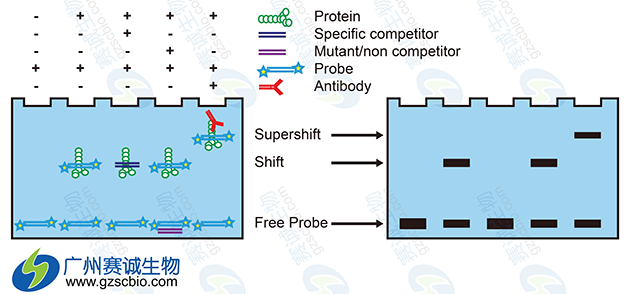
图5 EMSA实验原理
5 Luciferase实验
荧光素酶(Luciferase)是自然界中能够产生生物发光的酶的统称,不同的能够控制发光的生物体用不同的荧光素酶来催化不同的发光反应。
萤光生成反应通常分为以下两步:
萤光素+ ATP→ 萤光素化腺苷酸(luciferyl adenylate)+ PPi
萤光素化腺苷酸+ O2→ 氧萤光素+ AMP +光
荧光素酶的基因可以被合成并插入到生物体中或转染到细胞中,将不同类型的细胞(骨髓干细胞、T细胞等)标记上(即表达)荧光素酶,就可以用高敏感度的CCD相机进行对动物体内进行活体观察而不会伤害到动物本身。在荧光素酶中加入正确的萤光素底物就可以放出荧光,而发出的光子可以被光敏感元件,如萤光探测器或改进后的光学显微镜探测到。这就使得对包括感染在内的多种生命活动进程进行观察成为可能。
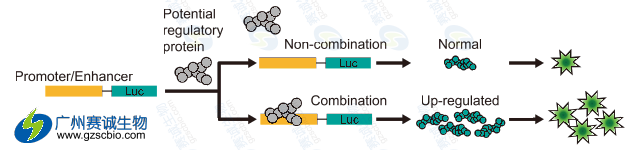
图6 Luciferase研究增强子原理
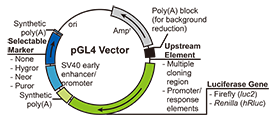
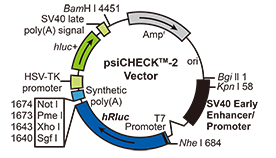
图7 Luciferase常用载体
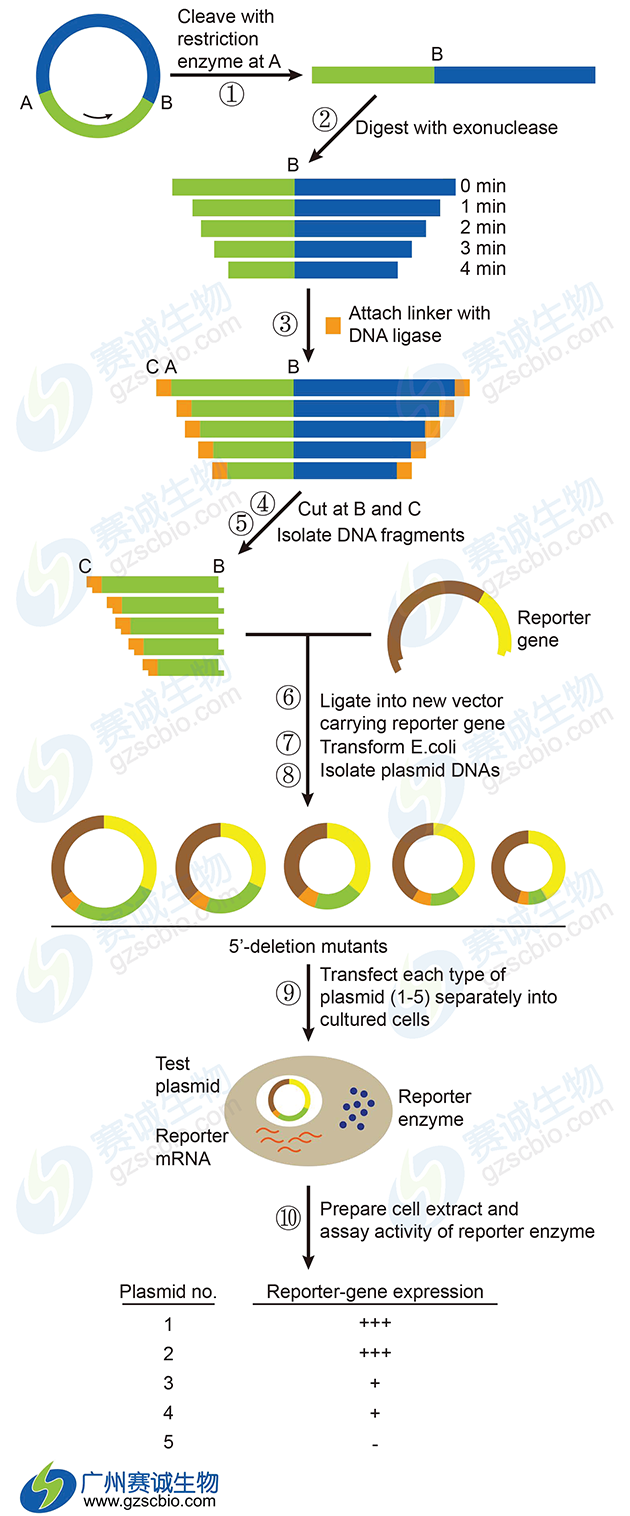
图8 Luciferase片段缺失分析实验流程
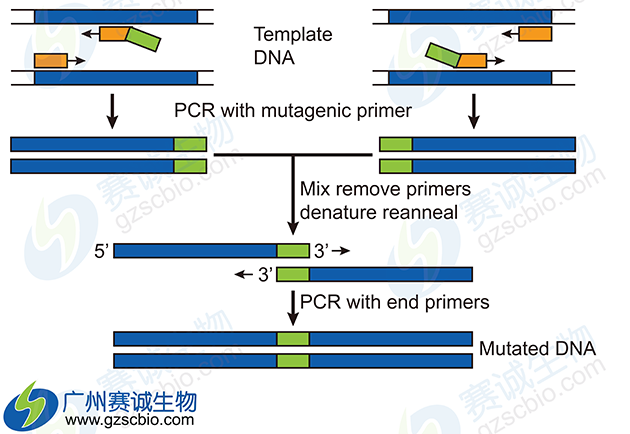
图9 Luciferase定点突变分析实验流程
荧光素酶分析可应用于启动子研究中分析顺式作用元件和反式作用因子、药物筛选、siRNA和miRNA筛选、分泌途径及蛋白定位报告基因检测、活细胞的实时动态研究、信号转导通路分析、难转染的细胞(包括干细胞和原代细胞)的研究、RNA剪接研究。
双荧光素酶报告基因测试,是结合萤火虫和海洋腔肠荧光素酶先进的共报告基因测试技术。在用萤火虫荧光素酶定量基因表达时,通常采用第二个报告基因来减少实验的变化因素。双荧光素酶报告基因测试系统,结合pRL载体系统,表达第二个报告基因海洋腔肠荧光素酶,在单管中进行双荧光素酶报告基因测试,快速、灵敏、简便。其应用广泛,可同时分析多个调控元件、同时分析多个信号转导通路、单次筛选一个以上的靶点(包括脱靶效应、分析两个或多个通路之间的相互作用)。